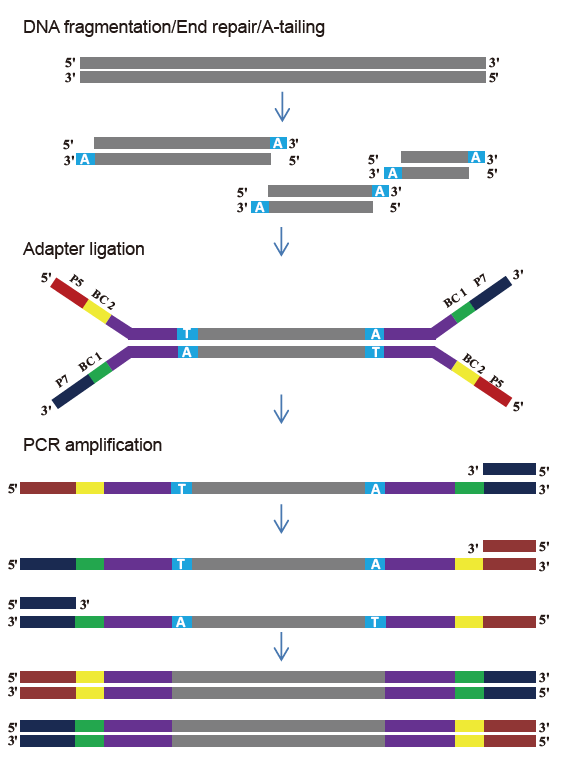
TIANSeq DirectFast Library Kit (belysning)
Funktioner
■ God ensartet sekventering: Ingen grundforskydning af DNA -fragmenteringsprocessen og PCR -amplifikationsprocessen.
■ Høj bibliotekskonverteringseffektivitet: Højeffektiv bibliotekskonstruktion kan sikres for 1 ng DNA-prøver.
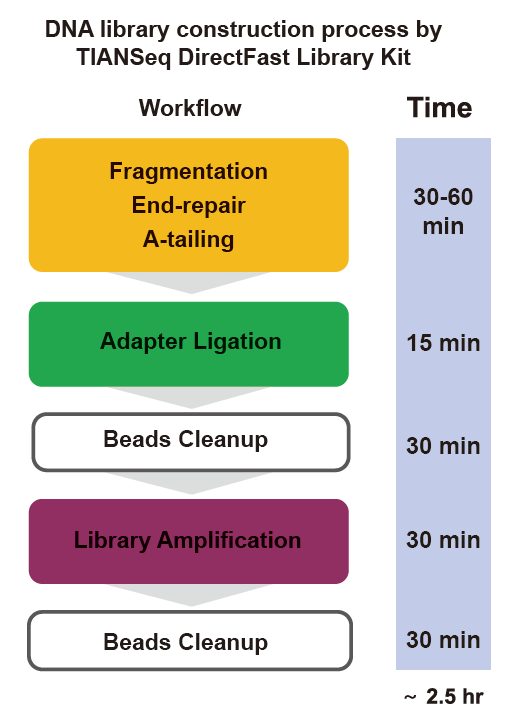
■ Hurtig drift: Hele bibliotekets byggeproces kræver kun 2,5 timer.
■ Omkostningseffektivt: Der kræves ingen særlige instrumenter og udstyr。
Specifikation
Type: DNA-biblioteksforberedelse til illumina high-throughput sekventeringsplatform
Prøve: Genomisk DNA eller stort fragment -DNA
Mål: Dobbeltstrenget DNA
Start prøveindgang: 1 ng- 1 μg
Driftstid: 2,5 time
Nedstrøms applikationer: Sekvensering på illumina platform
Alle produkterne kan tilpasses til ODM/OEM. For detaljer,klik venligst på Tilpasset service (ODM/OEM)
Fleksibel prøveindgang og fragmenteret størrelse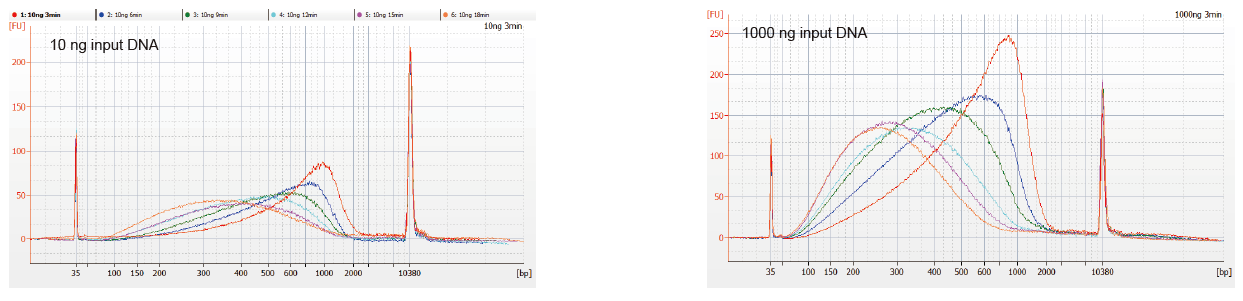 |
Figur 1. DNA -fragmenteringsprofiler med forskellig reaktionstid. 10 ng og 1000 ng DNA blev fragmenteret ved hjælp af TIANSeq DirectFast DNA Library Kit. Reaktionsprodukterne behandlet med forskellig reaktionstid blev oprenset med 1,8 × Ampure XP magnetiske perler og analyseret af Angilent 2100. |
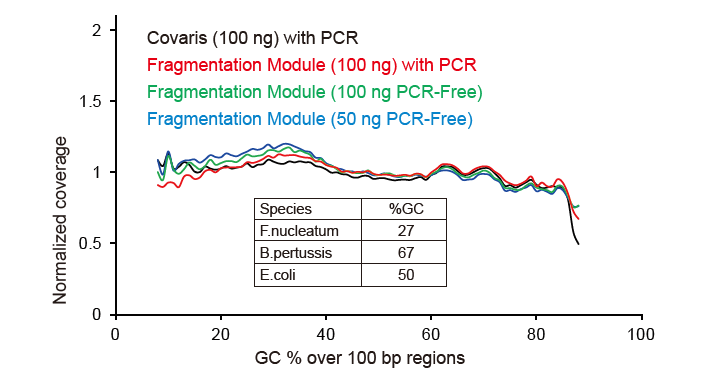
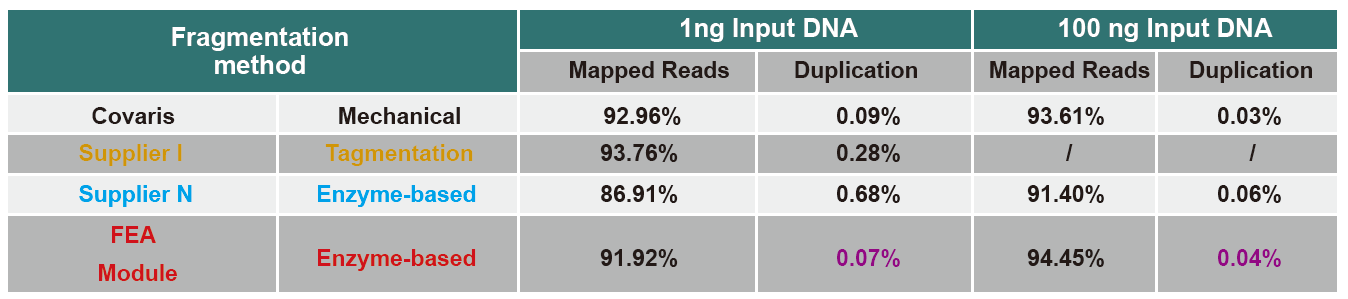
Covaris-lignende sekvensdækning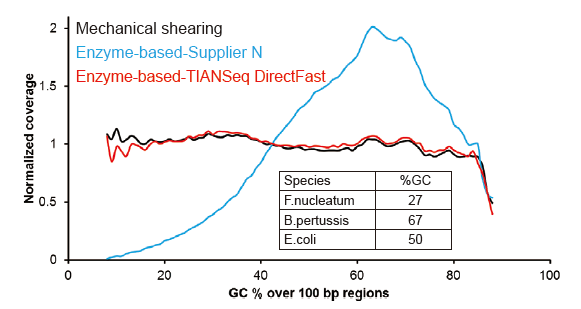 |
Figur 2. Sammenligning af genomdækning af forskellige biblioteksforberedelsesmetoder. Tre bakterielt genomisk DNA med forskelligt GC -indhold blandes ækvimolær, og sekvensering af genomdækningsresultat af 100 ng blandede DNA -biblioteker ved hjælp af disse metoder blev sammenlignet. Resultaterne viser, at TIANSeq DirectFast Library Kit har den samme effekt på DNA -fragmentering som mekanisk klipning, og der er ingen basisforskydning for fragmentering. |
Ingen systematisk bias for så lavt som 1 ng input -DNA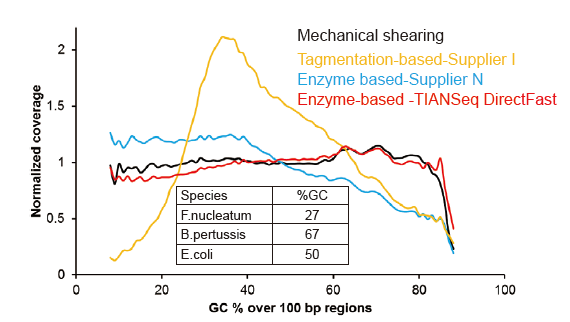 |
Figur 3. Sammenligning af genomdækning af forskellige biblioteksforberedelsesmetoder. Tre bakterielt genomisk DNA med forskelligt GC -indhold blandes ækvimolær, og sekvensering af genomdækningsresultat af 1 ng blandede DNA -biblioteker ved anvendelse af disse metoder blev sammenlignet. Resultaterne viser, at TIANSeq DirectFast Library Kit har en konsekvent fragmenteringseffekt med den mekaniske forskydning, selv for DNA -input så lavt som 1 ng, og der er ingen basisforskydning. |
| I stand til PCR-fri arbejdsgang
|
Figur 4. Forskellige input af genomisk DNA blev brugt til at konstruere biblioteket ved hjælp af PCR eller PCR-fri bibliotekskonstruktion, og genomets dækningsresultater blev sammenlignet. Resultaterne viser, at DNA-biblioteket, der er konstrueret med TIANSeq DirectFast Library Kit, ved hjælp af enrørsoperationen og effektive bibliotekskonstruktionstrinn opretholder en høj konsistens med den mekaniske forskydning i fragmentsekvensdækningsfordeling for både PCR-berigelse PCR-fri arbejdsgang. |
Statistik for bibliotekets konstruktionseffektivitet og udbytte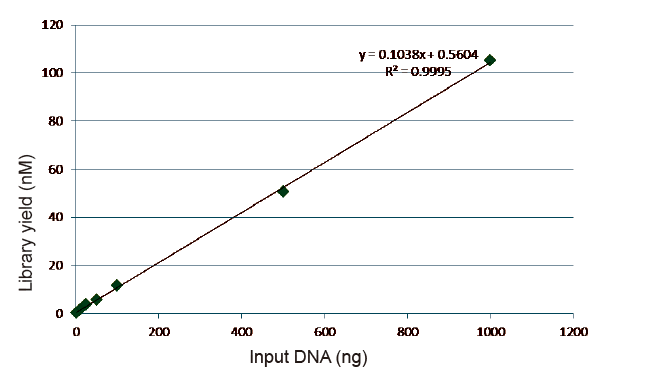 |
Figur 5. Resultater af kvantitativ analyse af biblioteks-DNA opnået ved qPCR efter bibliotekskonstruktion ved PCR-fri metode til prøver med forskellige startmængder (1, 10, 25, 50, 100, 500,1000 ng). Lineær regressionsanalyse viser, at biblioteksudbyttet har et godt lineært forhold i et bredt prøveindgangsområde. For DNA -input så lavt som 1 ng reduceres effektiviteten af bibliotekskonstruktion ikke. |
Sammenligning af sekventeringsdata for forskellige produkter
På nuværende tidspunkt er high-throughput sekventeringsteknologi hovedsageligt baseret på næste generations sekventeringsteknologi. Da læsningslængden af næste generations sekventeringsteknologi er begrænset, skal vi opdele sekvensen i fuld længde i små fragmentbiblioteker for at sekvensere. I henhold til behovene i forskellige sekventeringseksperimenter vælger vi normalt enkel sekvensering med én ende eller sekvensering med dobbelt ende. I øjeblikket er DNA-fragmenterne i den næste generations sekventeringsbibliotek generelt fordelt i området 200-800 bp.

a) DNA er af dårlig kvalitet og indeholder hæmmere. Brug DNA-prøver af høj kvalitet for at undgå hæmning af enzymaktivitet.
b) Mængden af DNA-prøve er utilstrækkelig ved anvendelse af PCR-fri metode til konstruktion af DNA-bibliotek. Når input af det fragmenterede DNA overstiger 50 ng, kan PCR-fri arbejdsgang udføres selektivt under bibliotekets konstruktionsproces. Hvis bibliotekets kopital er for lavt til at blive sekventeret direkte, kan DNA -biblioteket amplificeres ved PCR efter adapterligeringen.
c) RNA -kontaminering fører til unøjagtig initial DNA -kvantificering RNA -kontaminering kan eksistere i oprensningsprocessen for genomisk DNA, hvilket kan føre til unøjagtig DNA -kvantificering og utilstrækkelig DNA -belastning under bibliotekskonstruktion. RNA kan fjernes ved behandling med RNase.
A-1
a) Små fragmenter (60 bp-120 bp) vises Små fragmenter er normalt adapterfragmenter eller dimerer dannet af adaptere. Oprensning med Agencourt AMPure XP magnetiske perler kan effektivt fjerne disse adapterfragmenter og sikre sekventeringskvalitet.
b) Store fragmenter vises i biblioteket efter PCR -amplifikation Størrelsen af bibliotekets DNA -fragment vil stige med 120 bp, efter at adapteren er ligeret. Hvis DNA -fragmentet stiger med mere end 120 bp efter adapterligeringen, kan det skyldes unormal fragmentamplifikation af overdreven PCR -amplifikation. Reduktion af antallet af PCR -cyklusser kan forhindre situationen.
c) Unormal størrelse af bibliotek -DNA -fragmenter efter adapterligering Længden af adapteren i dette kit er 60 bp. Når fragmentets to ender ligeres til adapterne, vil længden kun stige med 120 bp. Når du bruger en anden adapter end den, der følger med dette kit, skal du kontakte leverandøren for at give relevante oplysninger, f.eks. Adapterlængde. Sørg for, at eksperimentets arbejdsgang og drift følger trinene beskrevet i manualen.
d) Unormal DNA -fragmentstørrelse før adapterligeringen Årsagen til dette problem kan skyldes forkerte reaktionsbetingelser under DNA -fragmentering. Forskellige reaktionstider bør bruges til forskellige DNA -input. Hvis DNA-input er mere end 10 ng, anbefaler vi at vælge reaktionstiden på 12 minutter som starttidspunkt for optimering, og fragmentstørrelsen, der produceres på dette tidspunkt, er hovedsageligt i området 300-500 bp. Brugere kan øge eller formindske længden af DNA-fragmenter i 2-4 minutter i henhold til deres egne krav for at optimere DNA-fragmenterne med den nødvendige størrelse.
A-2
a) Fragmenteringstiden er ikke optimeret Hvis det fragmenterede DNA er for lille eller for stort, se venligst retningslinjerne for valg af fragmenteringstid i instruktionen for at bestemme reaktionstiden, og brug dette tidspunkt som en kontrol, derudover opsæt en reaktionssystem for at forlænge eller forkorte 3 minutter for at foretage mere præcis justering af fragmenteringstiden.
A-3
Unormal størrelsesfordeling af DNA efter fragmenteringsbehandling
a) Forkert optøningsmetode for fragmenteringsreagens, eller hvis reagenset ikke blandes fuldstændigt efter optøning. Optø 5 × Fragmentation Enzyme Mix -reagenset på is. Efter optøning blandes reagenset jævnt ved forsigtigt at svinge i bunden af røret. Vend ikke reagenset!
b) DNA -inputprøven indeholder EDTA eller andre forurenende stoffer Nedbrydning af saltioner og chelateringsmidler i DNA -oprensningstrinnet er særlig vigtig for forsøgets succes. Hvis DNA er opløst i 1 × TE, skal du bruge metoden i instruktionen til at udføre fragmentering. Hvis EDTA -koncentrationen i opløsningen er usikker, anbefales det at rense DNA'et og opløse det i deioniseret vand til efterfølgende reaktion.
c) Unøjagtig initial DNA -kvantificering Størrelsen af fragmenteret DNA er tæt forbundet med mængden af DNA -input. Inden fragmenteringsbehandling er nøjagtig kvantificering af DNA ved hjælp af Qubit, Picogreen og andre metoder afgørende for at bestemme den nøjagtige mængde DNA i reaktionssystemet.
d) Fremstillingen af reaktionssystemet følger ikke instruktionerne. Fremstillingen af et fragmenteret reaktionssystem skal udføres på is strengt i henhold til instruktionerne. For at sikre den bedste effekt skal alle reaktionskomponenter placeres på is, og forberedelsen af reaktionssystemet skal udføres efter fuldstændig afkøling. Når forberedelsen er afsluttet, skal du svippe eller pipette for at blande grundigt. Vortex ikke!
1. Forkert blandingsmetode (vortex, voldsom svingning osv.) Vil forårsage unormal fordeling af biblioteksfragmenter (som vist i følgende figur) og dermed påvirke bibliotekets kvalitet. Derfor, når du forbereder Fragmentation Mix -reaktionsopløsningen, skal du forsigtigt pipette op og ned for at blande, eller brug fingerspidsen til at flikke og blande jævnt. Pas på ikke at blande med vortex.

2. DNA med høj renhed skal bruges til bibliotekskonstruktion
■ God DNA -integritet: Elektroforese -båndet er mere end 30 kb uden haler
■ OD260/230:> 1,5
■ OD260/280: 1,7-1,9
3. DNA -inputmængde skal være nøjagtig Det foreslås at bruge Qubit- og PicoGreen -metoder til at kvantificere DNA frem for Nanodrop.
4. Indholdet af EDTA i DNA -opløsning skal bestemmes EDTA har stor indflydelse på fragmenteringsreaktionen. Hvis indholdet af EDTA er højt, skal DNA -oprensning udføres før den efterfølgende test.
5. Fragmenteringsreaktionsopløsningen skal fremstilles på is. Fragmenteringsprocessen er følsom over for reaktionstemperatur og tid (især efter tilsætning af forstærker). For at sikre reaktionstidens nøjagtighed skal du forberede reaktionssystemet på is.
6. Fragmenteringsreaktionstiden skal være nøjagtig Reaktionstiden for fragmenteringstrinnet vil direkte påvirke fragmentprodukternes størrelse og dermed påvirke størrelsesfordelingen af DNA -fragmenter i biblioteket.
1. Hvilken type prøve er gældende for dette kit?
Den gældende prøvetype af dette kit kan være total RNA eller renset mRNA med god RNA -integritet. Hvis der bruges totalt RNA til at konstruere biblioteket, anbefales det at bruge rRNA -udtømningssættet (kat. 4992363/4992364/4992391) til først at fjerne rRNA.
2. Kan FFPE -prøver bruges til at konstruere bibliotek med dette kit?
MRNA i FFPE -prøver vil blive nedbrudt i et vist omfang med relativ dårlig integritet. Når du bruger dette kit til bibliotekskonstruktionen, anbefales det at optimere fragmenteringstiden (forkorte fragmenteringstiden eller ikke udføre fragmentering).
3. Hvad kan få det indsatte segment til at se en lille afvigelse ud ved hjælp af trinstørrelsesvalgstrinnet i produktmanualen?
Størrelsesvalg foretages i nøje overensstemmelse med størrelsesvalgstrinet i denne produktmanual. Hvis der er afvigelse, kan årsagen være, at de magnetiske perler ikke er afbalanceret til stuetemperatur eller ikke er fuldstændigt blandet, pipetten ikke er nøjagtig eller væsken forbliver i spidsen. Det anbefales at bruge tipene med lav adsorption til eksperimentet.
4. Valg af adaptere i bibliotekskonstruktion
Bibliotekets byggesæt indeholder ikke adapterreagens, og det anbefales at bruge dette kit sammen med TIANSeq Single-Index Adapter (Illumina) (4992641/4992642/4992378).
5. Bibliotekets QC
Bibliotekets kvantitative påvisning: Qubit og qPCR bruges til at bestemme henholdsvis massekoncentration og molkoncentration af biblioteket. Betjeningen er strengt i overensstemmelse med produktmanualen. Koncentrationen af biblioteket vil generelt opfylde kravene til NGS -sekventering. Registrering af biblioteksdistributionsområde: Brug af Agilent 2100 Bioanalyzer til at registrere bibliotekets distributionsområde.
6. Valg af forstærkningscyklusnummer
Ifølge instruktionerne er antallet af PCR-cyklusser 6-12, og antallet af nødvendige PCR-cykler skal vælges i henhold til prøveinput. I biblioteker med højt udbytte forekommer overforstærkning sædvanligvis i varierende grad, hvilket manifesteres af en lidt større top efter målintervallets top ved påvisning af Agilent 2100 Bioanalyzer, eller den påviste koncentration af Qubit er lavere end for qPCR. Mild over amplifikation er et normalt fænomen, som ikke påvirker bibliotekssekventering og efterfølgende dataanalyse.
7. Spikes vises i registreringsprofilen for Agilent 2100 Bioanalyzer
Udseendet af pigge ved Agilent 2100 Bioanalyzer -detektion skyldes den ujævne fragmentering af prøver, hvor der vil være flere fragmenter i en bestemt størrelse, og dette vil blive mere tydeligt efter PCR -berigelse. I dette tilfælde foreslås det ikke at udføre størrelsesudvælgelsen, dvs. indstille fragmenteringstilstanden til 94 ° C i 15 minutter inkuberet, hvor fragmentfordelingen er lille og koncentreret, og homogeniteten kan forbedres.
Produkter kategorier
HVORFOR VÆLGE OS
Siden etableringen har vores fabrik udviklet produkter i verdensklasse med overholdelse af princippet
af kvalitet først. Vores produkter har opnået et fremragende ry i branchen og er værdifulde blandt nye og gamle kunder.